Motif
Motif是DNA或RNA中存在的长度较短(6-8 bp),有特定模式的序列。 该序列一般具有一定的生物学功能,转录因子结合位点,RBP结合位点均憨厚特定的motif。 Motif的鉴定方法有: - 正则表达式 (Regular expression enumeration)
- 模式驱动
- K-mer+显著性检验
常用的工具有WEEDER(suffix tree implementation of RE motif hits) - 位置权重矩阵 (Position weigtht mstrix)
- EM算法
- Gibbs-sampling算法
- Greedy算法
mit-motif-course
Expectation Maximization for Motif Finding 算法
对象: - seq: 用于motif搜索的序列
- \(θ_0\): 没有motif存在的概率(基因组背景)
- θ: motif概率矩阵参数
- π: motif出现的位置
问题: P(θ, π | seq, \(θ_0\))最优化问题
方法: - π by P(π | θ, seq, \(θ_0\))
- θ by P(θ | π, seq, \(θ_0\))
- EM 和 Gibbs在估计的方法上存在差异
不同的初始化motif矩阵得到的结果不一样,所以MEME采用不同的kmer矩阵作为motif矩阵,计算每一个kmer,选出得分最高top100。
伪代码code 1
2
3
4
5
6
7
8# Basic EM Approach
given: length parameter W, training set of sequences
set initial values for p
do
re-estimate Z from p (E –step)
re-estimate p from Z (M-step)
until change in p < ε
return: p, Z 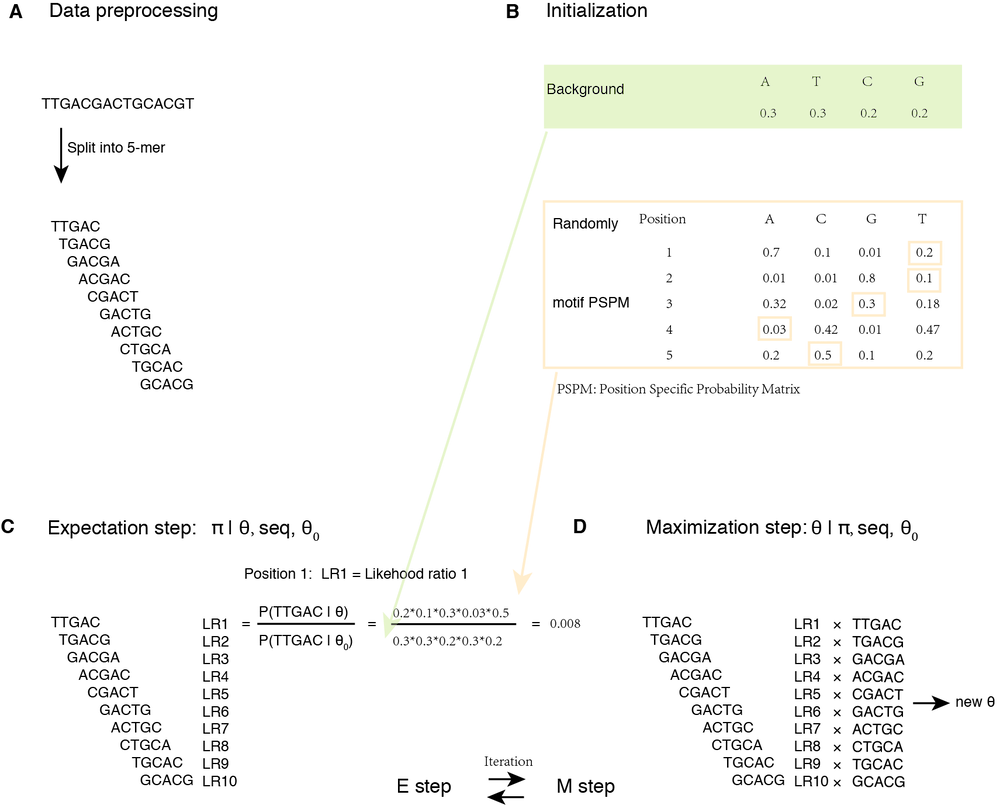
- Gibbs sampling Gibbs
1
2
3
4
5
6
7
8
9
10Gibbs Sampling Approach
given: length parameter W, training set of sequences
choose random positions for a
do
pick a sequence $X_i$
estimate p given current motif positions a (update step)
(using all sequences but $X_i$)
sample a new motif position $a_i$ for $X_i$ (sampling step)
until convergence
return: p, a